武汉金开瑞生物工程有限公司品牌商
12 年
手机商铺
- NaN
- 0.5
- 0.5
- 1.5
- 0.5
公司新闻/正文
收藏级!分子互作实验常用数据库、软件工具大合集(附链接)
759 人阅读发布时间:2025-06-27 10:03
在分子生物学的广袤领域中,深入理解分子间的相互作用对于揭示生命奥秘、攻克疾病难题以及开发创新疗法至关重要。今天,为大家精心整理了一份分子互作实验常用数据库及软件工具大合集,助力科研工作者在探索分子世界的道路上畅通无阻。
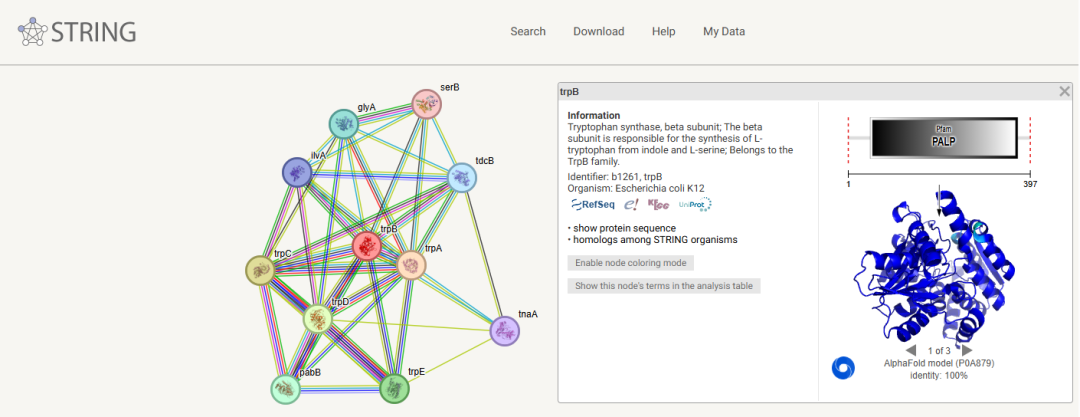
1. STRING
●简介:作为蛋白相互作用领域的老牌数据库,STRING 以其可靠的数据而闻名。它整合了多方面的数据来源,包括实验数据、从 PubMed 摘要中挖掘的结果、综合其他数据库数据以及生物信息学预测结果。该数据库覆盖物种极为广泛,互作信息丰富。
●使用方法:用户可输入蛋白质名称或序列(支持单个或多个蛋白)。输入基因名称后,能获得直观的蛋白互作网络图,图中圆圈节点间的直线代表蛋白间的相互作用关系,点击直线可查看详细信息。不同颜色直线对应不同相互作用关系证据来源,涵盖直接物理相互作用及间接功能相关性。
●网址:string - db.org/
2. IID
●简介:IID 是一个能够精确到器官特异性的蛋白相互作用数据库,收录了 18 个物种(含人类、5 个模式生物和 12 个驯化物种)中检测到和预测到的蛋白质 - 蛋白质相互作用(PPIs)。其为蛋白互作注释了全面信息,包括发生条件(如发育阶段、组织)、跨物种保护、方向性、可药用性、持续时间以及在多蛋白复合物中的相互作用情况。
●使用方法:通过特定的搜索界面,输入目标蛋白或物种相关信息,即可获取对应蛋白互作数据及详细注释。
●网址:iid.ophid.utoronto.ca/
3. BioGRID
●简介:BioGRID 是生物医学互动库,通过全面管理工作汇编数据。目前索引版本为 4.4.235,检索了大量出版物,涵盖众多蛋白质和遗传相互作用、化学相互作用及主要模式生物物种的翻译后修饰。所有数据免费提供搜索索引,并支持多种标准格式下载。
●使用方法:在搜索栏输入基因或蛋白名称,选择对应物种,提交搜索后可查看哪些基因具有相互作用,以及相互作用是基于实验还是预测。
●网址:thebiogrid.org/

1. JASPAR
●简介:JASPAR 是开源的转录因子结合位点信息数据库,以 position frequency matrices(PFMs)和 TF flexible models(TFFMs)形式记录了 6 大类物种转录因子的 DNA 结合偏好信息,这些信息可转换为位置权重矩阵(PWMs)用于扫描基因组序列。核心数据库 JASPAR CORE 涵盖脊椎动物、昆虫、线虫、真菌、植物和尾索动物等物种类群。
●使用方法:在数据库界面选择对应物种分类,输入相关转录因子或 DNA 序列信息,即可查询转录因子与 DNA 结合位点 motif 数据。
●网址:jaspar.elixir.no/
2. Harmonizonme
●简介:Harmonizonme 整合数十年研究成果,将基因和蛋白质信息从众多在线资源汇总到在线数据库。重点构建基因与基因、属性与属性的相似性网络,能直接提供靶基因数据,可在线浏览特定转录因子的靶基因,也方便下载数据集。
●使用方法:通过搜索功能,输入转录因子名称,即可获取其靶基因相关信息及下载链接。
●网址:maayanlab.cloud/Harmonizome/
3. TRRUST
●简介:TRRUST 是人工管理的人类和小鼠转录调控网络数据库。当前版本分别包含大量人类 TF 和小鼠 TF 的 TF - 靶标调控关系,这些关系来源于 pubmed 文章中的小规模实验研究。数据库运用文本挖掘方法从海量文章中搜索监管关系,并提供调节模式(激活或抑制)信息。
●使用方法:选择物种(人类或小鼠)后,输入转录因子名称,可查询转录因子对应的靶基因及转录因子间的调控关系和调节模式。
●网址:www.grnpedia.org/trrust/
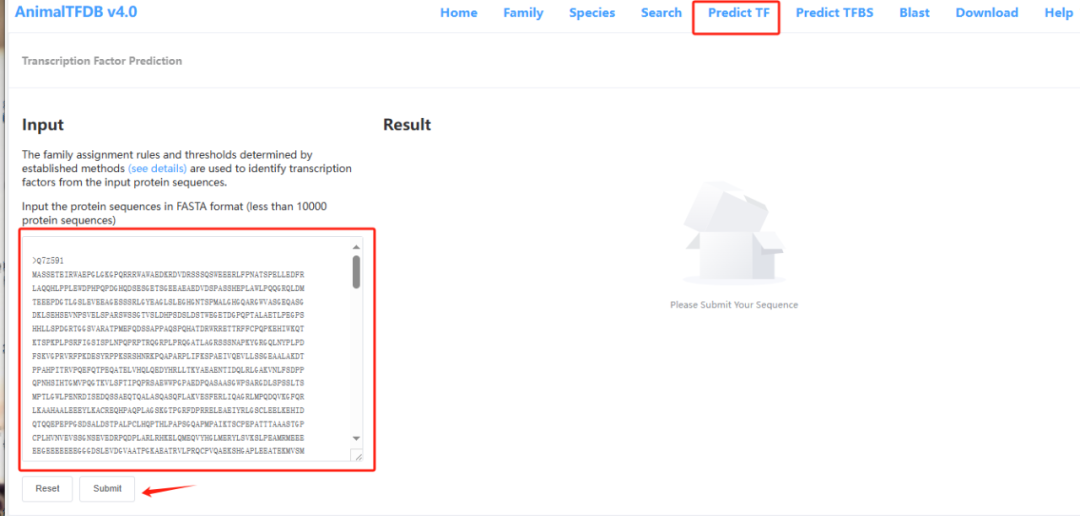
4.AnimalTFDB v4.0
●简介:AnimalTFDB v4.0 对 97 个动物基因组的转录因子(Transcription Factor)和转录辅助因子(Transcription cofactor)进行归纳整理。基于 DNA 结合结构域,将动物转录因子分成 73 个基因家族,转录辅助因子分成 83 个基因家族,并对它们进行了详细分类。
●使用方法:点击Predict TF跳转到转录因子预测页面,在文本框中输入fasta格式(fasta格式: 大于号 > 后紧跟序列名,换行后是序列)的蛋白序列信息(只支持蛋白序列文件),再点击Submit按钮即可将任务提交给后台运行,如果序列数据不多很快就可以反馈结果。
●网址:guolab.wchscu.cn/AnimalTFDB4/#/
5. hTFtarget
●简介:该数据库收录了多种细胞系、组织或细胞及干预因素条件下的大量实验样本大规模 ChIP - Seq 数据中 TFs 相关信息。其 TFs 靶基因数据基于 ChIP - Seq 数据分析和 TFBSs 分析两方面结果。
●使用方法:在数据库中输入转录因子信息,可通过转录因子预测靶基因。
●网址:guolab.wchscu.cn/hTFtarget/#!/search
1. catRAPID
●简介:catRAPID 是专门用于计算蛋白和 RNA 结合特性的工具,可从 RNA 或蛋白入手进行相应预测,在 lncRNA 研究等领域有重要应用。
●使用方法:在其操作界面,选择从 RNA 或蛋白方向进行分析,输入对应的 RNA 序列或蛋白信息,即可启动预测流程并获取结果。
●网址:service.tartaglialab.com/page/catrapid_group
2. StarBase v3.0
数据规模:整合 327 套 CLIP-seq 数据,覆盖 110 万 + RNA - 蛋白互作,支持人、鼠、果蝇等物种。
核心功能:
CLIP-seq 数据分析:通过整合 HITS-CLIP、PAR-CLIP 等数据,鉴定 RNA 结合蛋白(RBP)的结合位点。
泛癌症分析:基于 TCGA 数据,分析 RNA - 蛋白互作在 32 种癌症中的差异表达。
多组学整合:结合 RNA-seq、miRNA-seq 数据,预测 RBP 与疾病相关的体细胞突变关联。
点击网址:starbase.sysu.edu.cn/ ,进入官方主页可以看到:在主页面最上方,有序的排列着九个功能子菜单;中间部分安排着是对Starbase 数据库的介绍。
miRNA-Target:主要用来预测预测miRNA 的靶基因。提供 miRNA-mRNA、miRNA-lncRNA、miRNA-pseudogene、miRNA-circRNA 和 miRNA-sncRNA 五种的相互作用网络。
Degradome-RNA:提供了一种分析RNA 降解模式的综合方法。
RNA-RNA:主要进行全面预测候选ncRNA 及其靶标之间的 RNA-RNA 相互作用。
ceRNA-Network:包含3个子模块,即mRNA-ceRNA、lncRNA-ceRNA 和 pseudogene-ceRNA。分别用于通过竞争性结合 microRNA 来可视化 mRNA,lncRNA,转录的假基因及其亲本基因之间的串扰。

3. CircInteractome
网址:circinteractome.irp.nia.nih.gov/
数据规模:
预测 109 个 RBP 与 circRNA 的结合位点,覆盖 circBase 中所有 circRNA。基于 CLIP-seq 数据,识别 RBP 在 circRNA 反向剪切位点或侧翼序列的结合。提供 circRNA 特异性引物(Divergent Primers)和 siRNA 序列。

使用方法:
输入 circRNA 名称(如 “hsa_circ_0000234”)或宿主基因符号(如 “CUL2”)。
选择分析类型:
Circular RNA:查询 circRNA 基本信息及结合蛋白。
RBP on CircRNA:输入 RBP 名称(如 “HNRNPC”),检索与之结合的 circRNA。
结果页显示结合位点的基因组定位、RBP 功能注释及序列详情。
点击“Divergent Primers” 生成引物序列,用于 RT-PCR 验证。
4. RBPgroup
数据规模:整合 HEK293、HepG2 等细胞系的 327 套 CLIP-seq 数据,鉴定 RNA 调控元件。
通过非负矩阵分解(NMF)聚类 RBP,预测其组合调控模式。识别 RNA 靶标序列中的调控元件(如 miRNA 应答元件)。
使用方法:
上传 CLIP-seq 数据或输入基因名称(如 “TP53”)。
选择分析模式(如“RBP clustering” 或 “RNA motif enrichment”)。
结果页展示 RBP 聚类网络、调控元件序列及功能注释。
下载代码(GitHub)进行本地分析
网址:github.com/lulab/RBPgroup
1. miRanda
数据规模:预测 miRNA 与 mRNA、lncRNA 等的结合位点,覆盖人、鼠、果蝇等物种。
核心功能:
种子序列匹配:基于 miRNA 种子区(2-8 位)预测靶标。
多算法整合:结合 RNAhybrid、TargetScan 等工具提高准确性。
使用方法:
输入 miRNA 名称(如 “hsa-miR-34a”)和靶 RNA 序列(FASTA 格式)。
设置参数(如最小自由能阈值、种子匹配严格度)。
结果页展示结合位点的位置、热力学稳定性及保守性评分。
注册账号后可保存分析结果并跟踪更新。
网址:mirtoolsgallery.tech/mirtoolsgallery/node/1055
2. CircNet 2.0
数据规模重新整合了来自circAtlas和MiOncoCirc数据库的circRNA,以及来自从癌症基因组图谱(TCGA)数据库的新circRNA;重新整合了来自circAtlas和MiOncoCirc数据库的circRNA,以及来自从癌症基因组图谱(TCGA)数据库的新circRNA;、数据更准确,整合了来自miRTarBase数据库的384,897个经实验验证的miRNA-靶基因相互作用数据;数据更全面,新版增加了2,268 个癌症样本数据。
使用方法:
输入RNA 名称,选择RNA种类,点击search
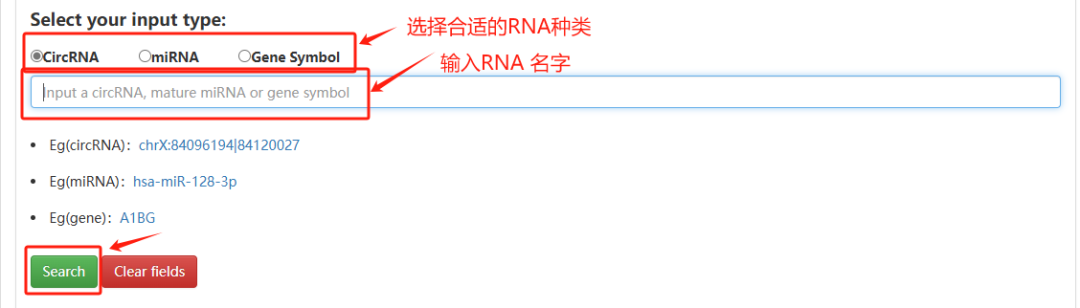
结果页展示互作网络拓扑图、表达量热图及功能富集分析。
点击“Export” 下载互作列表及序列文件。
网址:awi.cuhk.edu.cn/~CircNet/php/index.php